Hola,
BLAST+ es una de las pocas herramientas esenciales para cualquier persona que haga biología computacional, todos la hemos usado alguna vez. Simplificando, creo que el secreto es que hace una sola cosa (alinear localmente secuencias) y la hace bien. Eso, y que BLAST evoluciona, y de vez en cuando introduce novedades interesantes, que podemos repasar en las notas de publicación periódicas disponibles en https://www.ncbi.nlm.nih.gov/books/NBK131777
La versión 2.12.0 (28 de junio de 2021) incluye un nuevo modo de paralelización por lotes de secuencias input que beneficia entre otros a los algoritmos clásicos BLASTN, BLASTP y BLASTX. Los detalles puedes verlos en https://www.ncbi.nlm.nih.gov/books/NBK571452 , aquí comparto un ejemplo de invocación, que requiere el argumento -mt_mode 1:
$ blastp –db swissprot -query BIGFASTA.fsa –out test.out -num_threads 32 -mt_mode 1
La siguiente figura muestra la ganancia en tiempo de cálculo en función del número de cores y hebras empleado:
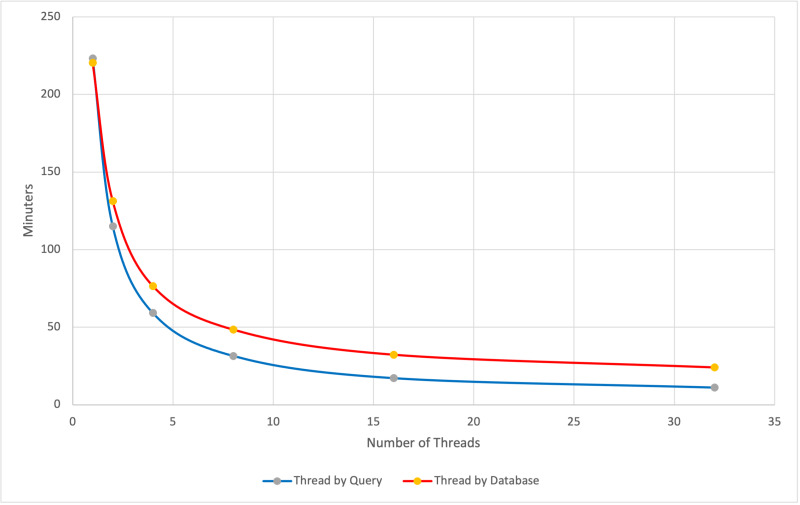
Espero que os sea útil, hasta pronto,
Bruno