La semana pasada anunciaron el premio Nobel de química de 2024:
- 1/2 a David Baker, “for computational protein design”
- 1/2 a Demis Hassabis y John M. Jumper “for protein structure prediction”
O como se ve en la figura, por jugar con proteínas en 3D:
| https://www.nobelprize.org/prizes/chemistry/2024/press-release |
Lo celebro en el blog porque hemos sido lectores de sus trabajos, usuarios de sus herramientas y divulgadores de sus avances desde hace años. Por temática, este premio sigue seguramente a éste otro. Hay muchas cosas que comentar de este premio, iré por partes. Si no conocéis el campo podéis revisar el material que durante años he ido revisando en http://eead-csic-compbio.github.io/bioinformatica_estructural .
En primer lugar, el grupo de David Baker ha sido un héroe del experimento CASP desde hace décadas y ya en 2011 le ponía como candidato a este premio. En la edición de 2003 (CASP5), en la que participé como parte de mi tesis, ya fueron los mejores en la categoría de nuevos plegamientos con el software Rosetta, por entonces escrito en FORTRAN. Para mi ha sido increíble ver cómo su grupo ha estado al frente todos estos años, incluso cuando AlphaFold (AF) les superó, ya en CASP13 y CASP14. A pesar de ello, nadie les ha superado diseñando proteínas y comprobando experimentalmente, por cristalografía y NMR, que funcionaban como esperaban. La suya ha sido sin duda una larga y exitosa carrera, de la que podéis leer más en inglés por ejemplo en el resumen de la Academia Sueca o en su lista de publicaciones.
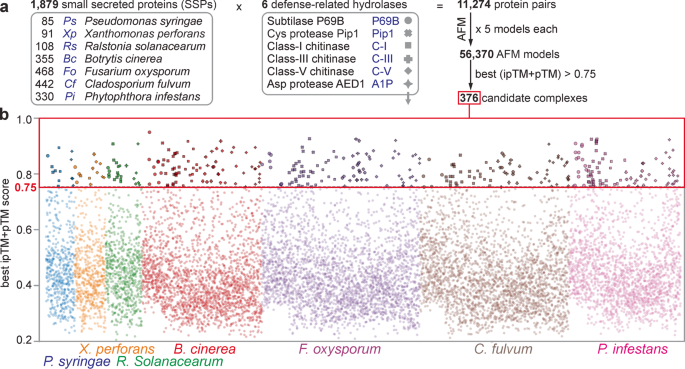
En segundo lugar, el premio se centra en el trabajo que llevó a producir el predictor AF2, del que hemos hablado largo y tendido en este blog, por ejemplo en la reciente entrada sobre un protocolo para modelar parejas de proteínas. En este caso los ganadores son fundadores y empleados de deepmind.google. A diferencia de David Baker llevan poco tiempo trabajando este área, pero han sabido mirar a hombros de gigantes de una manera muy eficiente, aprovechando lo aprendido por la comunidad CASP, la creciente colección de estructuras de alta calidad disponibles en el Protein Data Bank y las inabarcables bases de datos de secuencias naturales. Sin duda éste es un ejemplo más de un gran logro científico obtenido como resultado de muchos proyectos de ciencia básica y abierta, de la que Demis Hassabis es defensor, como nosotros en el blog. Es una pena que la versión AF3, que da menos peso a las secuencias naturales, no sea de momento de acceso abierto, algo que ha generado mucha polémica.
Hasta pronto,
Bruno
PD Si os gustan los hilos de Twitter podéis ver éste desde la perspectiva de Ewan Birney