Hola,
como ya hemos mencionado en otras ocasiones aquí, las proteínas habitualmente tienen uno o más dominios con determinadas funciones. Por eso cuando analizas secuencias de proteínas recursos como Pfam (incluída en Interpro) o CDD son muy útiles.
El crecimiento de las colecciones de secuencias es tan rápido que a veces se definen dominios o familias de proteínas sin saber realmente qué función tienen. Sabemos que existen, porque sus secuencias están conservadas en los genomas de diferentes organismos y se pueden alinear, pero todavía no hay evidencias de en qué procesos bioquímicos participan. Son los llamados Domains of Unknown Function (DUF).
Hace unos unos años Carlos Cantalapiedra y yo descubrimos tránscritos en cebada y en Arabidopsis thaliana que contenían dominios DUF. Entre ellos está por ejemplo DUF3615, pero todavía no sabemos si son importantes o no:
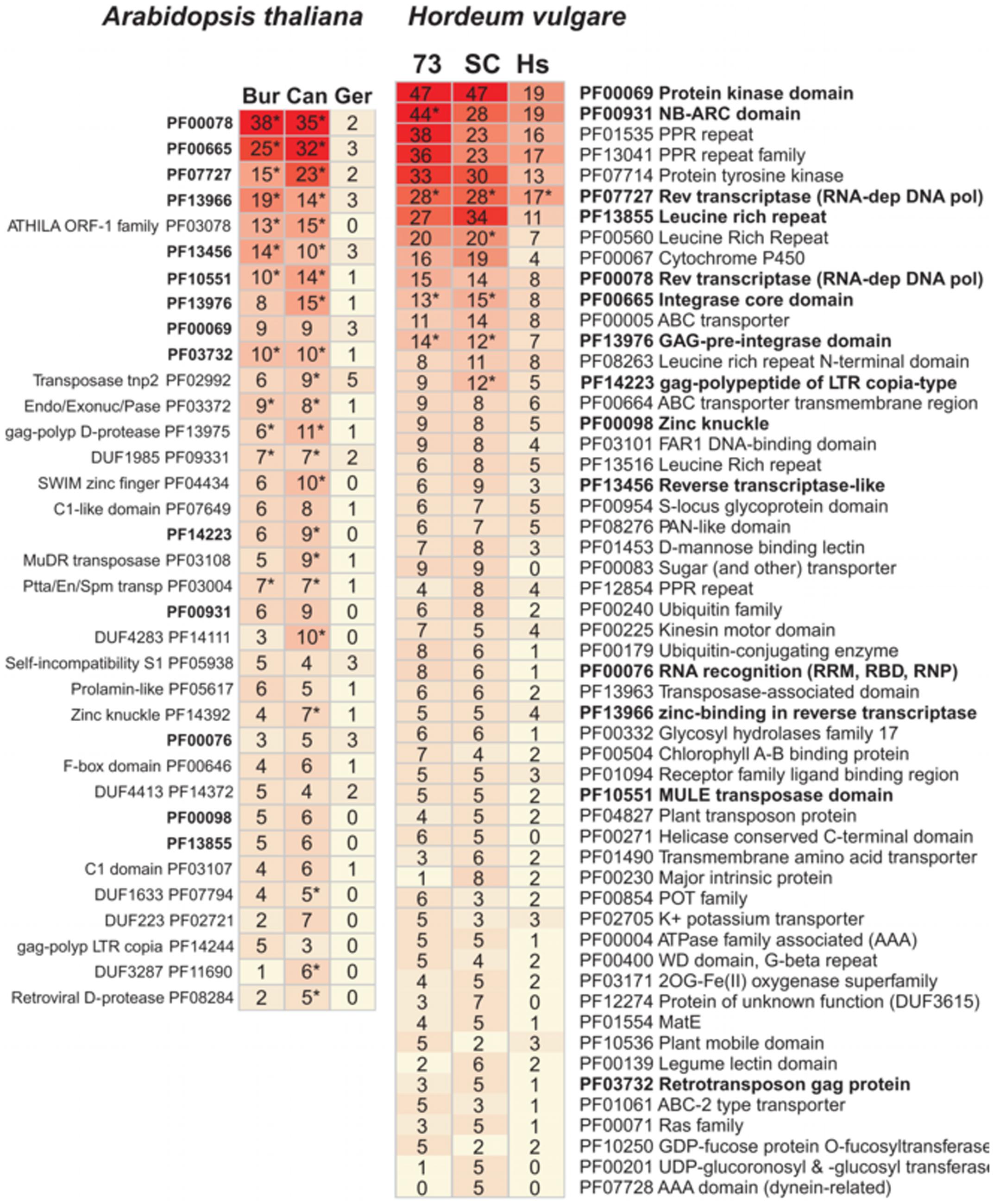 |
Figura 1. Dominios de Pfam encontrados en tránscritos accesorios de Arabidopsis thaliana (izq) y cebada (der). Fuente: https://doi.org/10.3389/fpls.2017.00184 |
La continuación de esta historia la encontramos en un artículo muy reciente, donde los autores descubren una pareja de proteínas, una de ellas DUF1644, que tanto en arroz como maiz interaccionan entre ellas y, al hacerlo, afectan al número de granos producidos, un caracter de enorme interés en la agricultura:
 |
| Figura 2. Interacción entre KRN2 y DUF1644 confirmada en ensayos Y1H (A) y ensayos de complementación con luciferasa en hojas de tabaco(B). Adaptada de https://doi.org/10.1126/science.abg7985 |
Queda claro que los dominios DUF son una fuente interesante por explorar. Lo lógico sería que con el tiempo se vayan convirtiendo en familias de función conocida, pero la verdad es que este ejemplo tampoco nos dice mucho de la función de DUF1644, solamente que interacciona con otras proteínas.
Hasta pronto,
Bruno