today I was visiting Rothamsted Research invited to the Brassica's BRAVO stakeholders meeting. There I could listen to SteveDorling (University East Anglia) talk about the UK Climate Projections 2018. These are my notes on that.
He basically talked us about the challenges of choosing the right climate dataset for each agriculture problem (https://www.metoffice.gov.uk/research/collaboration/ukcp/download-data , https://www.metoffice.gov.uk/climate/uk/data/haduk-grid/haduk-grid) and in other resources such as http://www.cordex.org. The UKCP2018 data are available for the period 1900-2100. Regional projections are currently available with 12km resolution, and later in the year with a 2.2km grid assuming the current rate of greenhouse emissions. The headline for the UK is that as temperatures rise, winter precipitation will increase as much as they will be reduced in the summer.
Which variables will likely change most so that they can be considered in future Brassica experiments? That depends on the resolution and timescales, and whether we are talking of UK field trials or seed production overseas, but Steve singled out “chilling”, soil temperature or soil moisture. He added that suitability models are promising tools, such as https://www.tandfonline.com/doi/full/10.1080/1747423X.2018.1537312.
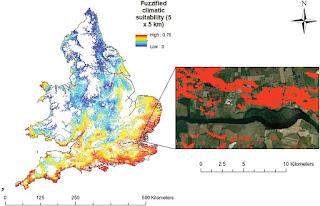 |
| Source: https://www.tandfonline.com/doi/full/10.1080/1747423X.2018.1537312 |
Note that in this example they used a 5x5km grid, just like our recent work on "Genetic association with high‐resolution climate data reveals selection footprints in the genomes of barley landraces across the Iberian Peninsula" (see https://doi.org/10.1111/mec.15009).
He believes that the current climate datasets are so large that they should support any question we might come up with with help from Machine Learning,
Bruno



