# Obtains the format of a file
sub obtain_file_format {
my ($file) = @_;
open(FILE,$file)|| die "# $0 : cannot read $file\n";
my %score;
while(<FILE>){
if (/^#/){
next;
}
if (/^>/){
$score{'fasta'}++;
}
if (/^(LOCUS|DEFINITION|ACCESSION|FEATURES)/){
$score{'genbank'}++;
}
if (/^(HEADER|TITLE|COMPND|SEQRES|ATOM)/){
$score{'pdb'}++;
}
if (/^(DE|VV|XX)/){
$score{'transfac'}++;
}
}
close(FILE);
my @sorted_scores;
if (@sorted_scores = sort { $score{$b} <=> $score{$a} } (keys %score)) {
return $sorted_scores[0];
} else {
return 'unknown';
}
}
Ideas y código para problemas de genómica de plantas, biología computacional y estructural
6 de agosto de 2010
Script para conocer el formato de un archivo
En el campo de la bioinformática se trabaja con numerosos formatos de archivos de datos biológicos (FASTA, GenBank, PDB...). Habitualmente tenemos que conocer el formato de los datos de un archivo que usamos como entrada en un script. Sin embargo, esto no es tarea fácil puesto que diferentes formatos pueden contener el mismo tipo de datos de forma poco estandarizada. En estos casos tenemos que usar nuestros conocimientos previos sobre el formato para establecer el tipo de archivo. El siguiente código de perl permite introducir información sobre los formatos más habituales en forma de expresiones regulares para posteriormente leer un fichero y asignar una puntuación a cada uno de los formatos definidos. De acuerdo a la puntuación final conoceremos el formato de fichero más probable.
30 de julio de 2010
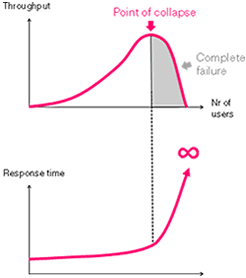
Bioinformática en la nube a partir de 10000 usuarios
La comunidad bioinformática tiene ya una cierta tradición en poner a disposición de los usuarios el software desarrollado a través de la red. Esto sirve para dar a conocer las últimas ideas y algoritmos que se van desarrollando en los laboratorios, de manera que se ponen a prueba en escenarios reales en manos de usuarios que no son sus creadores. Por supuesto, también sirve para ir ampliando el repertorio de herramientas que cualquiera con conexión a Internet puede usar, normalmente de forma gratuita.
Para el usuario final éstas son a menudo simplemente aplicaciones accesibles desde un navegador web, pero para sus creadores son programas más o menos complejos que viven en servidores web (como máquinas virtuales) que consumen luz y hay que administrar, que son a veces objeto de ataques informáticos, y que dependiendo del uso que tengan exigirán ampliaciones de hardware.
Qué coste real tienen estos servidores? El coste absoluto de mantener un servidor es difícil de promediar, porque dependerá de costes (luz, hardware, la carga de usuarios) y beneficios (evaluación peer to peer de los algoritmos, corrección de errores, retroalimentación de los usuarios, incluso citas bibliográficas) variables.
Otra cuestión es si es mejor alojar estos servidores en nuestras propias máquinas o aprovecharnos de las arquitecturas de computación en la nube, que son escalables. Para ayudarnos a tomar esta decisión un reciente artículo en Bioinformatics del grupo de Cedric Notredame, donde prueban la versión paralelizada de su famoso alineador múltiple Tcoffee (basado en el principio de consistencia), sugiere que con los costes actuales sólo compensa alojar este tipo de servicios web en arquitecturas en nube a partir de 10000 usuarios al mes.
Nos vemos a la vuelta de las vacaciones.
Para el usuario final éstas son a menudo simplemente aplicaciones accesibles desde un navegador web, pero para sus creadores son programas más o menos complejos que viven en servidores web (como máquinas virtuales) que consumen luz y hay que administrar, que son a veces objeto de ataques informáticos, y que dependiendo del uso que tengan exigirán ampliaciones de hardware.
Qué coste real tienen estos servidores? El coste absoluto de mantener un servidor es difícil de promediar, porque dependerá de costes (luz, hardware, la carga de usuarios) y beneficios (evaluación peer to peer de los algoritmos, corrección de errores, retroalimentación de los usuarios, incluso citas bibliográficas) variables.
Otra cuestión es si es mejor alojar estos servidores en nuestras propias máquinas o aprovecharnos de las arquitecturas de computación en la nube, que son escalables. Para ayudarnos a tomar esta decisión un reciente artículo en Bioinformatics del grupo de Cedric Notredame, donde prueban la versión paralelizada de su famoso alineador múltiple Tcoffee (basado en el principio de consistencia), sugiere que con los costes actuales sólo compensa alojar este tipo de servicios web en arquitecturas en nube a partir de 10000 usuarios al mes.
Nos vemos a la vuelta de las vacaciones.
19 de julio de 2010
Alineamiento perfil-perfil teniendo en cuenta la estructura secundaria
Con el alineamiento múltiple de secuencias de proteínas relacionadas se puede construir un perfil. A parte de su uso en técnicas de fold recognition o para predicción de estructura secundaria, este se puede utilizar para compararlo con las secuencias de otras proteínas e incluso con otro grupo de proteínas relacionadas entre sí, en este caso mediante una comparación perfil-perfil.
En el siguiente ejemplo, escrito en python y del que hay un ejemplo en perl aquí, vamos a realizar la comparación entre 2 perfiles, y apoyaremos el alineamiento con la información disponible de la estructura secundaria predicha para ambos. Utilizaremos un algoritmo de programación dinámica, por lo que si no se está familiarizado con la técnica se puede acudir a uno de los muchos libros sobre algoritmos o al excelente artículo de Sean R Eddy What is dynamic programming?, el cual se basa precisamente en ejemplos de alineamiento de secuencias biológicas.
Básicamente, necesitaremos un archivo PSSM y otro PSIPRED, perfil y estructura secundaria, respectivamente, para cada uno de los 2 grupos de proteínas. Puedes encontrar archivos de ejemplo en este curso, donde ya enlazamos para el ejemplo en perl.
Remarcar, entre los aspectos más importantes del código, que la puntuación entre perfiles se calcula como la media entre los valores de las 2 matrices y que se asigna una puntuación extra al caso de que 2 posiciones alineadas tengan el mismo estado de estructura secundaria.
Esta sería la salida del programa, donde es importante fijarse en el alineamiento obtenido (que se muestra aquí en 2 fragmentos por motivo de espacio) y cómo, a pesar de que pocos aminoácidos coinciden, los estados de estructura secundaria se muestran alineados, lo que podría interpretarse como que la estructura está más conservada que la secuencia.
En el siguiente ejemplo, escrito en python y del que hay un ejemplo en perl aquí, vamos a realizar la comparación entre 2 perfiles, y apoyaremos el alineamiento con la información disponible de la estructura secundaria predicha para ambos. Utilizaremos un algoritmo de programación dinámica, por lo que si no se está familiarizado con la técnica se puede acudir a uno de los muchos libros sobre algoritmos o al excelente artículo de Sean R Eddy What is dynamic programming?, el cual se basa precisamente en ejemplos de alineamiento de secuencias biológicas.
Básicamente, necesitaremos un archivo PSSM y otro PSIPRED, perfil y estructura secundaria, respectivamente, para cada uno de los 2 grupos de proteínas. Puedes encontrar archivos de ejemplo en este curso, donde ya enlazamos para el ejemplo en perl.
Remarcar, entre los aspectos más importantes del código, que la puntuación entre perfiles se calcula como la media entre los valores de las 2 matrices y que se asigna una puntuación extra al caso de que 2 posiciones alineadas tengan el mismo estado de estructura secundaria.
#!/usr/bin/python
from __future__ import with_statement
import re
__title__ = "Secondary structure and pairwise DP profile-profile alignment"
__author__ = "Carlos P Cantalapiedra"
__institution__ = "CSIC AulaDei Laboratory of Computational Biology"
### MAIN STARTS UNDERNEATH THE FUNCTIONS ###
# Function that parses PSSM files
# The format of the array is:
# pssm[
# {'aa':aa1, 'weights', [w1, w2, ...]},
# {'aa':aa2, 'weights', [w1, w2, ...]},
# .
# .
# .
# ]
def read_PSSM(pssm_file):
pssm = [] # Returning dictionary
aa = ''
weights = []
# Finds the aminoacid (\w) and his weights ((\s*-?\d*.\d*)+)
reg_exp = re.compile(r"^\s*\d+\s+(\w)((\s*-?\d*.\d*)+)")
# NOTE: with the with_statement there's no need to close the file
with open(pssm_file, 'r') as file:
for line in file:
result = reg_exp.search(line)
if result:
try:
aa = result.group(1)
weights = str(result.group(2)).split()
except IndexError:
raise IOError(\
"There is a problem with the format of the PSSM file "+str(pssm_file))
pssm.append({'aa' : aa, 'weights' : weights})
return pssm
# Function that parses "Pred:" line of PSIPRED files,
# to obtain the predicted secondary structures
def read_PSIPRED(psipred_file):
psipred = []
reg_exp = re.compile(r"^\s*Pred:\s*(\w+)")
# NOTE: with the with_statement there's no need to close the file
with open(psipred_file, 'r') as file:
for line in file:
result = reg_exp.search(line)
if result:
try:
[psipred.append(a) for a in result.group(1)]
except IndexError:
raise IOError(\
"There is a problem with the format of the PSIPRED file "+str(psipred_file))
return psipred
# Function that returns the bonus score associated to secondary structure
def get_secondary_score(st1, st2):
global secondary_score
score = 0
if st1 == st2 and st2 != 'C':
score = secondary_score
return score
# Function that creates de Dynamic Programming matrix
# The format of the matrix is going to be:
# matrix[
# row(0) [('r', gap), ('r', gap), ..., ('r', gap)] # len(row) = len(prot2) + 1
# row(1) [('c', gap), (path, score), ..., (path,score)]
# row(2) [('c', gap), (path, score), ..., (path,score)]
# .
# .
# .
# row(len(prot1)) [('c', gap), (path, score), ..., (path, score)]
# # len(col) = len(prot1) + 1
# ]
#
def create_matrix_DP(pssm1, pssm2, psipred1, psipred2):
global aa_order
global gapcost
matrix_DP = [] # Returning matrix
scores = []
score = 0
score1 = 0 # score1 and score2 serve several purposes
score2 = 0
aa1_count = 0 # 2 indexes
aa2_count = 0
path = '' # 'd': diagonal, 'c': column, 'r': row
try:
# Fisrt row is set up with gap values
init_row = [('r', i * gapcost) for i in range(0, len(psipred2) + 1)]
matrix_DP.append(init_row)
for aa1 in pssm1:
aa1_count+=1
index1 = aa_order.index(aa1['aa']) # aa1 position in PSSM columns
scores = []
aa2_count = 0
for aa2 in pssm2:
aa2_count+=1
# First column is set up with gap values
if aa2_count == 1:
scores.append(('c', aa1_count * gapcost))
index2 = aa_order.index(aa2['aa'])
score1 = int(aa2['weights'][index1])
score2 = int(aa1['weights'][index2])
score = (score1 + score2) / 2.0
score += get_secondary_score(psipred1[aa1_count-1], psipred2[aa2_count-1])
# Now does apply the function for matrix positions:
# Sij = max (score(-1,-1)+score, score(-1,0)+gap, score(0,-1)+gap)
# [1] is the score in the tuple (path, score)
score += matrix_DP[aa1_count - 1][aa2_count - 1][1]
score_1 = matrix_DP[aa1_count - 1][aa2_count][1] + gapcost
score_2 = scores[aa2_count - 1][1] + gapcost
if score_1 >= score and score_1 >= score_2:
path = 'c' # Column
score = score_1
elif score_2 >= score:
path = 'r' # Row
score = score_2
else:
path = 'd' # Diagonal
# Adds the score to the list of current row
scores.append((path, score))
# Adds a row of scores
matrix_DP.append(scores)
except ValueError, value:
raise Exception("Error. Maybe there is a rare aminoacid in the PSSM.\n"+value)
except IndexError, value:
raise Exception("Error. "+str(value))
return matrix_DP
# Recursive function that recovers the alignment
def visit_alignment(i, j, output):
global matrix_DP # This could be changed to be a parameter by reference
global prot1_pssm, prot2_pssm, prot1_psipred, prot2_psipred
if i == 0 and j == 0: return # END OF RECURSIVE SEARCH
join = ' '
if prot1_pssm[i - 1]['aa'] == prot2_pssm[j - 1]['aa']: join = '|'
path = matrix_DP[i][j][0]
score = matrix_DP[i][j][1]
if path == 'd':
visit_alignment(i - 1, j - 1, output)
output.append((prot1_psipred[i - 1], prot1_pssm[i - 1]['aa'],\
join, prot2_pssm[j - 1]['aa'], prot2_psipred[j - 1]))
elif path == 'c':
visit_alignment(i - 1, j, output)
output.append((prot1_psipred[i - 1], prot1_pssm[i - 1]['aa'], join, '-', '-'))
elif path == 'r':
visit_alignment(i, j - 1, output)
output.append(('-', '-', join, prot2_pssm[j - 1]['aa'], prot2_psipred[j - 1]))
else:
raise Exception("Invalid path "+path)
return
############################ MAIN ##################################
# Global variables: program parameters
aa_order = ('ARNDCQEGHILKMFPSTWYV'); # This must copy aa order in PSSM columns
gapcost = -8
secondary_score = 2
# File locations: please, change this variables to suit your needs
prot1_pssm_file = '../pssm/1ngk_A.pssm'
prot2_pssm_file = '../pssm/1s69_A.pssm'
prot1_psipred_file = '../psipred/1ngk_A.psipred'
prot2_psipred_file = '../psipred/1s69_A.psipred'
# Printing title and author
print
print "*** "+__title__+" ***"
print "Author: "+__author__+" at "+__institution__
# Printing parameters
print
print "Aminoacids and order (PSSM files): "+aa_order
print "Gap penalty = "+str(gapcost)
print "Equal secondary structure score = "+str(secondary_score)
### 1) Opening and parsing PSSM and PSIPRED files
prot1_pssm = []
prot2_pssm = []
prot1_psipred = []
prot2_psipred = []
try:
print
prot1_pssm = read_PSSM(prot1_pssm_file)
prot2_pssm = read_PSSM(prot2_pssm_file)
print "> PSSM files successfully opened."
prot1_psipred = read_PSIPRED(prot1_psipred_file)
prot2_psipred = read_PSIPRED(prot2_psipred_file)
print "> PSIPRED files successfully opened."
except IOError, value:
print "There has been an error reading input files:"
print value
### 2) Creating the matrix of DP
matrix_DP = [[]]
len_prot1 = len(prot1_psipred)
len_prot2 = len(prot2_psipred)
try:
matrix_DP = create_matrix_DP(prot1_pssm, prot2_pssm, prot1_psipred, prot2_psipred)
except Exception, value:
print value
### 3) Printing the maximum score
print
print "Maximum score "+str(matrix_DP[len_prot1][len_prot2][1])
### 4) Recovering and printing the alignment
output = [] # [(ss1, aa1, align, aa2, ss2)]
# Lines to print
ss1 = ''
aa1 = ''
align = ''
aa2 = ''
ss2 = ''
try:
# Recursively recovering the alignment
visit_alignment(len_prot1, len_prot2, output)
# Printing the alignment
print
for nts in output:
ss1 += str(nts[0])
aa1 += str(nts[1])
align += str(nts[2])
aa2 += str(nts[3])
ss2 += str(nts[4])
print ss1
print aa1
print align
print aa2
print ss2
except Exception, value:
print "Error while tracing the alignment."
print value
else: # END OF PROGRAM
print
print "*** Alignment finished successfully ***"
Esta sería la salida del programa, donde es importante fijarse en el alineamiento obtenido (que se muestra aquí en 2 fragmentos por motivo de espacio) y cómo, a pesar de que pocos aminoácidos coinciden, los estados de estructura secundaria se muestran alineados, lo que podría interpretarse como que la estructura está más conservada que la secuencia.
*** Secondary structure and pairwise DP profile-profile alignment ***
Author: Carlos P Cantalapiedra at CSIC AulaDei Laboratory of Computational Biology
Aminoacids and order (PSSM files): ARNDCQEGHILKMFPSTWYV
Gap penalty = -8
Equal secondary structure score = 2
> PSSM files successfully opened.
> PSIPRED files successfully opened.
Maximum score 444.0
CCHHHHHCCHHHHHHHHHHHHHHHHCCHHHHHHCCCCCHHHHHHHHHHHHHHHHCCCCCCCCCCCCCCH
KSFYDAVGGAKTFDAIVSRFYAQVAEDEVLRRVYPEDDLAGAEERLRMFLEQYWGGPRTYSEQRGHPRL
| || | | || | | | | || || |
STLYEKLGGTTAVDLAVDKFYERVLQDDRIKHFFADVDMAKQRAHQKAFLTYAFGGTDKYDGRYMREAH
CCHHHHHCCHHHHHHHHHHHHHHHHCCHHHHHHCCCCCHHHHHHHHHHHHHHHHCCCCCCCCCCHHHHH
HHCCCCCCCCHHHHHHHHHHHHHHHHHHCCCCCCHHHHHHHHHHHHHHHH--HHHCCCC
RMRHAPFRISLIERDAWLRCMHTAVASIDSETLDDEHRRELLDYLEMAAH--SLVNSPF
|| | || |
KELVENHGLNGEHFDAVAEDLLATLKEMG---VPEDLIAEVAAVAGAPAHKRDVLNQ--
HCCCCCCCCCHHHHHHHHHHHHHHHHHCC---CCHHHHHHHHHHHHHHHHHHHHHCC--
*** Alignment finished successfully ***
13 de julio de 2010
Eliminar secuencias redundantes y transcritos repetidos (II, con CD-HIT)
Como complemento a la entrada anterior de Álvaro os paso un enlace al programa
CD-HIT, que es una opción muy cómoda y eficiente para eliminar redundancia dentro de un conjunto de secuencias de proteínas o de DNA. El artículo donde se describe la primera versión de CD-HIT se publicó en Bioinformatics en 2006.
Aunque hay una serie de servidores web que pueden ayudarnos en esta tarea, si tienes un volumen realmente grande de secuencias lo más probable es que necesites descargar el código fuente (en C++) y compilarlo con make. Una vez compilado, el programa tiene múltiples opciones, pero su uso es muy sencillo. El algoritmo se basa en calcular la identidad de todas las secuencias (previamente ordenadas por longitud) por parejas para ir generando clusters de secuencias con una identidad superior al umbral deseado. Por defecto el umbral de identidad es del 90%, medida a lo largo de toda la secuencia (global), no sólo la alineada. Por supuesto se puede modificar este comportamiento y elegir por ejemplo umbrales más bajos o identidades locales.
Terminaré con un ejemplo de uso. Por ejemplo, dado el mismo archivo FASTA de la entrada anterior de Álvaro, para obtener el subconjunto no redundante al 80% sería necesario el siguiente comando en el terminal Linux:
$ cd-hit/cd-hit -i redundante.faa -o noredundante.faa -c 0.8
Como resultado, además del fichero noredundante.faa, se obtienen otros dos archivos (noredundante.faa.clstr y noredundante.faa.bak.clstr) que contienen
los clusters encontrados y las secuencias redundantes observadas.
CD-HIT, que es una opción muy cómoda y eficiente para eliminar redundancia dentro de un conjunto de secuencias de proteínas o de DNA. El artículo donde se describe la primera versión de CD-HIT se publicó en Bioinformatics en 2006.
Aunque hay una serie de servidores web que pueden ayudarnos en esta tarea, si tienes un volumen realmente grande de secuencias lo más probable es que necesites descargar el código fuente (en C++) y compilarlo con make. Una vez compilado, el programa tiene múltiples opciones, pero su uso es muy sencillo. El algoritmo se basa en calcular la identidad de todas las secuencias (previamente ordenadas por longitud) por parejas para ir generando clusters de secuencias con una identidad superior al umbral deseado. Por defecto el umbral de identidad es del 90%, medida a lo largo de toda la secuencia (global), no sólo la alineada. Por supuesto se puede modificar este comportamiento y elegir por ejemplo umbrales más bajos o identidades locales.
Terminaré con un ejemplo de uso. Por ejemplo, dado el mismo archivo FASTA de la entrada anterior de Álvaro, para obtener el subconjunto no redundante al 80% sería necesario el siguiente comando en el terminal Linux:
$ cd-hit/cd-hit -i redundante.faa -o noredundante.faa -c 0.8
Como resultado, además del fichero noredundante.faa, se obtienen otros dos archivos (noredundante.faa.clstr y noredundante.faa.bak.clstr) que contienen
los clusters encontrados y las secuencias redundantes observadas.
5 de julio de 2010
Eliminar secuencias redundantes y transcritos repetidos en genomas y proteomas
Una situación cada vez más habitual en genómica y proteómica es la redundancia de datos. Con las modernas técnicas de secuenciación de alto rendimiento se generan gigas y gigas de datos que es necesario filtrar y corregir para obtener resultados inteligibles.
El problema más habitual y sencillo es la redundancia de secuencias, cuando en un mismo fichero aparecen secuencias idénticas con identificadores diferentes. En otras ocasiones el problema es el contrario, identificadores idénticos apuntan a secuencias parciales de una misma secuencia (especialmente en datos de proteómica o transcriptómica).
El siguiente script filtra un fichero de sequencias en formato FASTA, dos tipos de filtro son posibles: 1) filtrar las secuencias redundantes (repetidas), 2) filtrar las secuencias de transcritos parciales (recuperando únicamente los transcritos de mayor secuencia en ficheros de proteínas)
An example FASTA file to test the script:
El problema más habitual y sencillo es la redundancia de secuencias, cuando en un mismo fichero aparecen secuencias idénticas con identificadores diferentes. En otras ocasiones el problema es el contrario, identificadores idénticos apuntan a secuencias parciales de una misma secuencia (especialmente en datos de proteómica o transcriptómica).
El siguiente script filtra un fichero de sequencias en formato FASTA, dos tipos de filtro son posibles: 1) filtrar las secuencias redundantes (repetidas), 2) filtrar las secuencias de transcritos parciales (recuperando únicamente los transcritos de mayor secuencia en ficheros de proteínas)
#!/usr/bin/perl
my ($infile) = @ARGV;
my $outfile = filter_fasta_file($infile,['longtrans','nonredundant']);
# Create a FASTA file with custom filtered sequences
sub filter_fasta_file {
my ($infile, $filter, $outfile) = @_;
if (!defined($outfile)){$outfile=$infile.'.filtered'}
open(INFILE,"$infile");
open(OUTFILE,">$outfile");
my ($sequences,$names,$headers) = read_FASTA_file($infile);
my (@previous_sequences,@previous_names,@previous_headers);
# my (@filtered_sequences,@filtered_names,@fitered_headers);
for (my $i=0; $i<=$#{$headers}; $i++){
my $write=1;
if (in_array($filter,'nonredundant')){
my ($found,$posic)=in_array($sequences, $sequences->[$i], 1);
foreach my $pos (@{$posic}){
if ($i != $pos && $i > $pos){
$write = 0; last;
}
}
}
if (in_array($filter,'longtrans')){
$names->[$i] =~ /([^\.]+)\.?(\d?)/;
my $name = $1;
my $variant = $2;
my ($found,$posic)=in_array($names, "/$1/", 1);
foreach my $pos (@{$posic}){
if ($i != $pos){
if (length($sequences->[$i]) < length($sequences->[$pos])) {
$write = 0; last;
}
if (length($sequences->[$i]) == length($sequences->[$pos]) && $sequences->[$i] eq $sequences->[$pos]){
$names->[$pos] =~ /([^\.]+)\.?(\d?)/;
my $name2 = $1;
my $variant2 = $2;
if ($variant2 < $variant){
$write = 0; last;
}
}
}
}
}
if ($write) {
print OUTFILE $headers->[$i]."\n".$sequences->[$i]."\n";
}
}
close INFILE;
close OUTFILE;
return $outfile;
}
#################################################################################
# Create 3 array references with sequences, names and headers from a FASTA file
sub read_FASTA_file
{
my($FASTAfile,$full_header,$allow_empty_sequences,$allow_multi_rows) = @_;
if(!$full_header){ $full_header = $allow_empty_sequences = 0 }
if(!$allow_empty_sequences){ $allow_empty_sequences = 0 }
if(!$allow_multi_rows){ $allow_multi_rows = 0 }
my (@sequences,@names,@headers,$seq,$seqname,$header);
my $headerOK = 0;
open(FASTA,$FASTAfile) || die "# read_FASTA_file : cannot find $FASTAfile\n";
$seq = '';
while(<FASTA>){
next if(/^#/); # allow comments
s/\012\015?|\015\012?|^\s+|\s+$//;
if(/>/){
$headerOK++;
if($seq || ($allow_empty_sequences && $seqname)){
if(!$allow_multi_rows){
$seq =~ s/\d|\s//g;
}
push(@sequences,$seq); # store this FASTA sequence
push(@names,$seqname);
push(@headers,$header);
$seq='';
}
if($full_header){
$seqname = substr($_,1);
}else { # get next sequence name
if (/^>\s*([^\t|^\||^;]+)/){
$header = $_;
$seqname = $1;
if (length($seqname)>32 && $seqname =~ /^([^\s]+)+\s/){
$seqname = $1;
}
$seqname =~ s/^\s+|\s+$// ;
}
}
}else{
if(!$allow_multi_rows){
s/\s+//g;
}
$seq .= $_;
}
}
close(FASTA);
# take care of last FASTA sequence
push(@sequences,$seq);
push(@names,$seqname);
push(@headers,$header);
if(!$headerOK){ @sequences = @names = @headers = (); } #Aug2007
return (\@sequences,\@names,\@headers);
}
#################################################################################
# Searchs a string inside an array
sub in_array {
my ($array,$search_for,$search_posic) = @_;
# my %items = map {$_ => 1} @$array; # create a hash out of the array values
# my $exist = (exists($items{$search_for}))?1:0;
# return (exists($items{$search_for}))?1:0;
my @posic;
# If search a pattern
if ($search_for =~ /^\/.+\/$/){
$search_for =~ s/^\/|\/$//g;
@posic = grep { $array->[$_] =~ /$search_for/ } 0 .. $#{$array};
} else {
@posic = grep { $array->[$_] eq $search_for } 0 .. $#{$array};
}
if (defined($search_posic) && $search_posic){
(scalar @posic)?return (1,\@posic):return (0);
} else {
(scalar @posic)?return 1:return 0;
}
}
An example FASTA file to test the script:
>AT1G51370.2 | Symbols: | F-box family protein | chr1:19045615-19046748 FORWARD
MVGGKKKTKICDKVSHEEDRISQLPEPLISEILFHLSTKDSVRTSALSTKWRYLWQSVPG
LDLDPYASSNTNTIVSFVESFFDSHRDSWIRKLRLDLGYHHDKYDLMSWIDAATTRRIQH
LDVHCFHDNKIPLSIYTCTTLVHLRLRWAVLTNPEFVSLPCLKIMHFENVSYPNETTLQK
LISGSPVLEELILFSTMYPKGNVLQLRSDTLKRLDINEFIDVVIYAPLLQCLRAKMYSTK
NFQIISSGFPAKLDIDFVNTGGRYQKKKVIEDILIDISRVRDLVISSNTWKEFFLYSKSR
PLLQFRYISHLNARFYISDLEMLPTLLESCPKLESLILVMSSFNPS*
>AT1G70790.1 | Symbols: | C2 domain-containing protein | chr1:26700724-26702127 FORWARD
MEDKPLGILRVHVKRGINLAIRDATTSDPYVVITLANQKLKTRVINNNCNPVWNEQLTLS
IKDVNDPIRLTVFDKDRFSGDDKMGDAEIDFRPFLEAHQMELDFQKLPNGCAIKRIRPGR
TNCLAEESSITWSNGKIMQEMILRLKNVECGEVELMLEWTDGPGCKGLGREGSKKTPWMP
TKRLD*
>AT1G50920.1 | Symbols: | GTP-binding protein-related | chr1:18870555-18872570 FORWARD
MVQYNFKRITVVPNGKEFVDIILSRTQRQTPTVVHKGYKINRLRQFYMRKVKYTQTNFHA
KLSAIIDEFPRLEQIHPFYGDLLHVLYNKDHYKLALGQVNTARNLISKISKDYVKLLKYG
DSLYRCKCLKVAALGRMCTVLKRITPSLAYLEQIRQHMARLPSIDPNTRTVLICGYPNVG
KSSFMNKVTRADVDVQPYAFTTKSLFVGHTDYKYLRYQVIDTPGILDRPFEDRNIIEMCS
ITALAHLRAAVLFFLDISGSCGYTIAQQAALFHSIKSLFMNKPLVIVCNKTDLMPMENIS
EEDRKLIEEMKSEAMKTAMGASEEQVLLKMSTLTDEGVMSVKNAACERLLDQRVEAKMKS
KKINDHLNRFHVAIPKPRDSIERLPCIPQVVLEAKAKEAAAMEKRKTEKDLEEENGGAGV
YSASLKKNYILQHDEWKEDIMPEILDGHNVADFIDPDILQRLAELEREEGIREAGVEEAD
MEMDIEKLSDEQLKQLSEIRKKKAILIKNHRLKKTVAQNRSTVPRKFDKDKKYTTKRMGR
ELSAMGLDPSSAMDRARSKSRGRKRDRSEDAGNDAMDVDDEQQSNKKQRVRSKSRAMSIS
RSQSRPPAHEVVPGEGFKDSTQKLSAIKISNKSHKKRDKNARRGEADRVIPTLRPKHLFS
GKRGKGKTDRR*
>AT1G70795.1 | Symbols: | C2 domain-containing protein | chr1:26700724-26702127 FORWARD
MEDKPLGILRVHVKRGINLAIRDATTSDPYVVITLANQKLKTRVINNNCNPVWNEQLTLS
IKDVNDPIRLTVFDKDRFSGDDKMGDAEIDFRPFLEAHQMELDFQKLPNGCAIKRIRPGR
TNCLAEESSITWSNGKIMQEMILRLKNVECGEVELMLEWTDGPGCKGLGREGSKKTPWMP
TKRLD*
>AT1G70790.2 | Symbols: | C2 domain-containing protein | chr1:26700724-26702127 FORWARD
MEDKPLGILRVHVKRGINLAIRDATTSDPYVVITLANQKLKTRVINNNCNPVWNEQLTLS
IKDVNDPIRLTVFDKDRFSGDDKMGDAEIDFRPFLEAHQMELDFQKLPNGCAIKRIRPGR
TNCLAEESSITWSNGKIMQEMILRLKNVECGEVELMLEWTDGPGCKGLGREGSKKTPWMP
TKRLD*
>AT1G36960.1 | Symbols: | unknown protein | chr1:14014796-14015508 FORWARD
MTRLLPYKGGDFLGPDFLTFIDLCVQVRGIPLPYLSELTVSFIAGTLGPILEMEFNQDTS
TYVAFIRVKIRLVFIDRLRFFRREEAAASNTITDQTHMTSSNSSDISPASPISQPPLPAS
LPSHDSYFDAGIQASRLVNPRAISQHHFSSSYSDFKGKEKAKIKIGECSKRKKDKQVDSG
T*
>AT1G51370.1 | Symbols: | F-box family protein | chr1:19045615-19047141 FORWARD
MVGGKKKTKICDKVSHEEDRISQLPEPLISEILFHLSTKDSVRTSALSTKWRYLWQSVPG
LDLDPYASSNTNTIVSFVESFFDSHRDSWIRKLRLDLGYHHDKYDLMSWIDAATTRRIQH
LDVHCFHDNKIPLSIYTCTTLVHLRLRWAVLTNPEFVSLPCLKIMHFENVSYPNETTLQK
LISGSPVLEELILFSTMYPKGNVLQLRSDTLKRLDINEFIDVVIYAPLLQCLRAKMYSTK
NFQIISSGFPAKLDIDFVNTGGRYQKKKVIEDILIDISRVRDLVISSNTWKEFFLYSKSR
PLLQFRYISHLNARFYISDLEMLPTLLESCPKLESLILEMVKNQSTRRHGEKREPNVMVS
TVPWCLVSSLKFVELKRSIPRYEGEMELVRYVLTNSTVLKKLRLNVYYTKKAKCAFLTEL
VAIPRCSSTCVVLVL*
Suscribirse a:
Entradas (Atom)