Hola, una tarea frecuente en mejora de plantas es obtener secuencias genómicas de un panel de individuos, por ejemplo variedades de melocotonero o de cebada, para mapear zonas del genoma asociadas a caracteres de interés (GWAS).
Una manera efectiva de hacerlo es secuenciar solamente una fracción del genoma, una que maximice la información obtenida, para reducir costes. Najla Ksouri, una joven investigadora de nuestro campus, acaba de publicar un protocolo para esta tarea. En este caso se basa en la metodología ddRAD-seq, que secuencia fragmentos de ADN obtenidos tras digerir ADN nuclear con una pareja de enzimas de restricción elegidas para representar todo el genoma y minimizar las regiones repetidas.
El protocolo está disponible en https://github.com/najlaksouri/GWAS-Workflow y comprende las siguientes etapas, que a su vez requieren de scripts en el repositorio: 
El protocolo usa como ejemplo datos de melocotonero, publicados en https://doi.org/10.1186/s13007-025-01415-3, pero en principio se puede aplicar a otras especies diploides sin modificaciones.
La siguiente figura muestra un ejemplo de picos significativos obtenidos por GWAS en varios cromosomas, en este caso para fecha de cosecha:
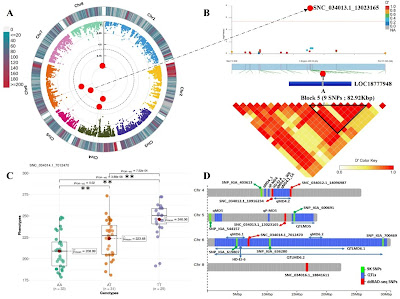 |
| Ejemplo de picos de GWAS (A), bloque ligado (B), carácter asociado (C) y genes candidato (D), tomado de https://doi.org/10.1186/s13007-025-01415-3 |
Un saludo
