Hola,
la pandemia que estamos viviendo, un año después, nos está
poniendo a prueba. A pesar de la improvisación de los políticos a escala global, de la desinformación en las redes sociales y los brotes de desconfianza, a pesar de la economía bajo mínimos y del cole en casa, ahora la mayoría tenemos la esperanza de que las vacunas resuelvan el problema.
En este artículo solamente pretendo recordar el largo camino de la investigación básica que nos ha traído hasta el presente. Estas vacunas son ya grandes hitos de la humanidad, a la altura de la llegada a la luna, pero el camino ha sido largo, de al menos 25 años. Por tanto, nada de milagros, son el fruto de mucho trabajo acumulado que fue explotado con mucho éxito por empresas como BioNTech y Moderna. Como pasó con CRISPR para la edicion de genomas, para que alguien llegara a la cima fueron necesarios muchos pasos previos, muchos de los cuales fuera de contexto serían objeto de "y eso para qué sirve". Aquí enumero los más importantes para las vacunas de ARN, extraídos de este hilo (no están los de los otros tipos de vacunas):
1970 T7 ARN polimerasa: https://nature.com/articles/228227a0 . Esta enzima permite sintetizar moléculas de ARN a medida, como las de las vacunas.
1978 Liposomas para llevar ARN mensajeros (mRNA): https://nature.com/articles/274923a0 . Estos vehículos permiten que el ARN de las vacunas pueda atravesar
la doble capa lipídica de la membrana celular.
1990 Inyecciones de ADN y ARN para expresar genes de manera transitoria en tejidos: https://science.sciencemag.org/content/247/4949/1465.long . Este trabajo demostró que es posible expresar genes a medida tras ser inyectados en tejidos, de manera que se traducen como proteínas.
2005 Ribonucleótidos modificados no disparan respuestas inmunes: https://cell.com/immunity/fulltext/S1074-7613(05)00211-6 .
Esto permite que el ARN inyectado no desencadene una reacción inmune por si mismo, lo que se pretende es que la reacción la desencadene la proteína codificada por ese ARNm.
2017 Estabilización de proteínas expuestas de los coronavirus MERS-CoV y SARS-CoV: https://pnas.org/content/114/35. Esto permite que la proteína modificada del coronoavirus que expresa el ARNm sea más estable y desencadene una reacción inmune más robusta.
Esta sucesión de descubrimientos y la tecnología actual permitieron que el tiempo de desarrollo de las vacunas haya sido el más corto de la historia:
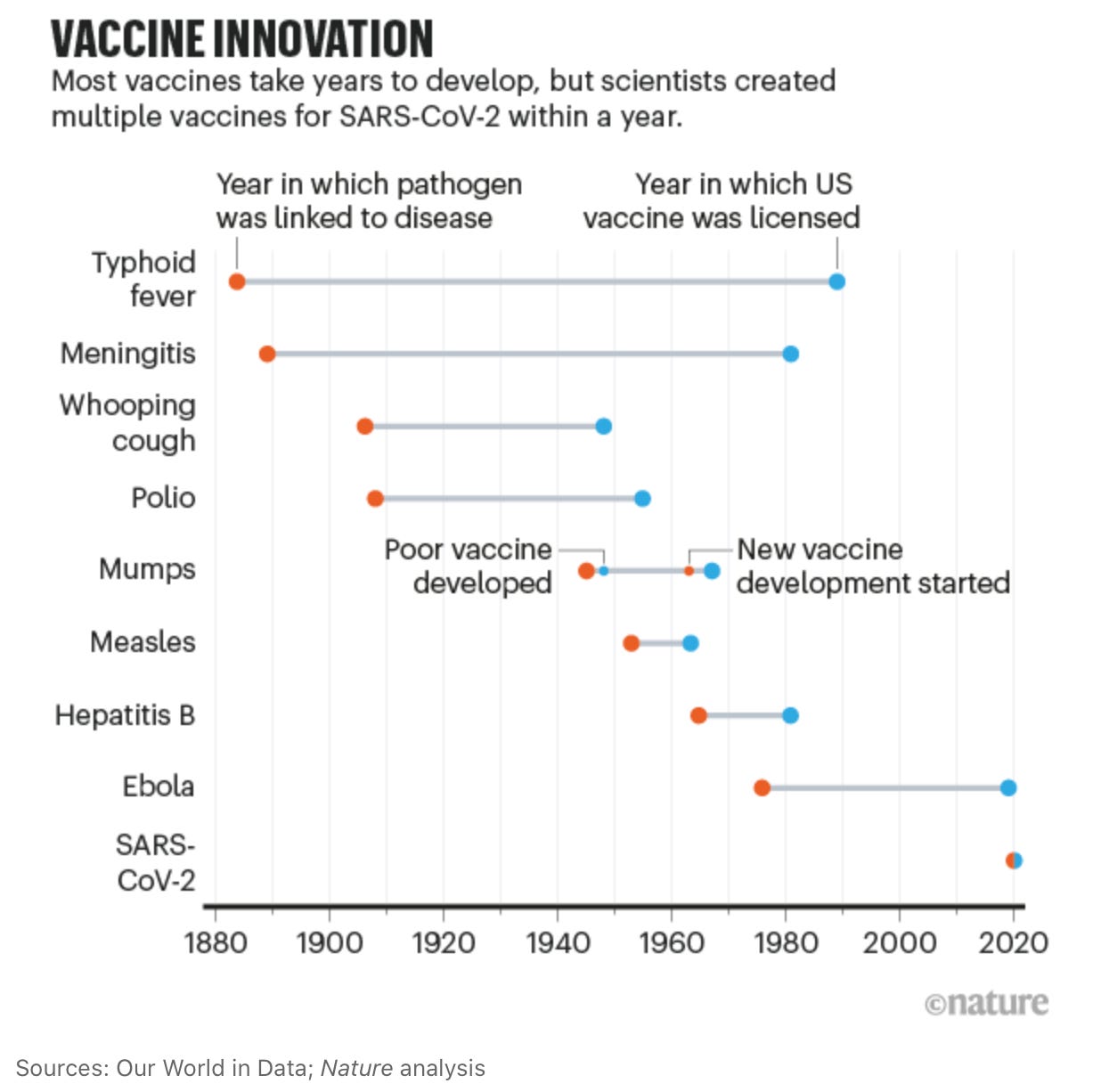
En mi actual institución, el Instituto Europeo de Bioinformática (EBI), también hemos contribuído con el https://www.covid19dataportal.org y el navegador de genomas https://covid-19.ensembl.org
Espero haberos convencido de que la ciencia financiada con fondos públicos y transparente, muchas veces invisible e ingrata, es una parte fundamental e integral del avance de nuestra sociedad, y que de ella beben las empresas que nos venden luego los productos.
Hasta pronto,
Bruno
PD1 Me preguntan si "mucha de esa investigación no la financian las farmacéuticas de forma privada, con escasos beneficios en mucho casos. Esto me recuerda a cuando proponen nacionalizar farmacéuticas".
Mi respuesta es que mi argumento no iba en esa dirección. De hecho creo que esta historia es un buen ejemplo de cómo lo iniciativa privada puede agilizar el desarrollo y las pruebas clínicas de las vacunas llevando a buen puerto en tiempo récord lo que empezó solamente como investigación básica. Mi argumento es que el riesgo es que la opinión pública, y los políticos que deciden, se queden sólo con el final del proceso y no vea la utilidad de la investigación básica. Quién les iba a decir a los autores de los artículos citados que iban a ser instrumentales para dos vacunas en el año 2020?
En cuanto a la financiación, como no es mi campo no me atrevo a opinar, pero sí puedo copiar aquí los que dicen al respecto los artículos citados más arriba:
1970 "This investigation was supported by a US Public Health Service research grant and training grant from the Institute of General Medical Sciences."
1978 "I thank Dr J. R. Tata for the freedom to pursue my own research goals... G.J.D. is in receipt of an EMBO long-term fellowship."
1990 "Supported in part by the NIH (grant numbers HD00669-05 and HD03352) and the Lynn F. Taylor Memorial Fund."
2005 "This work was supported by National Institutes of Health grants AI060505, AI50484, and DE14825."
2017 "This work was supported
by Grants P20GM113132 ... and R01AI127521 ..., NIH Contract HHSN261200800001E Agreement 6x142 ..., and intramural funding from National Institute of Allergy and
Infectious Diseases to support work at the VRC. Argonne is
operated by UChicago Argonne, LLC, for the US Department of Energy
(DOE), Office of Biological and Environmental Research under Contract
DE-AC02-06CH11357. Use of the Stanford Synchrotron Radiation Lightsource
(SSRL), SLAC National Accelerator Laboratory, is supported by the DOE,
Office of Science, Office of Basic Energy Sciences under Contract
DE-AC02-76SF00515. The SSRL Structural Molecular Biology Program is
supported by the DOE Office of Biological and Environmental Research and
by the NIH, National Institute of General Medical Sciences (including
P41GM103393)."
PD2 Parece ser que Uğur Şahin, uno de los dos fundadores de BioNTech, dirige actualmente un proyecto ERC financiado con dinero público de la UE: https://twitter.com/ERC_Research/status/1372962936856190982

