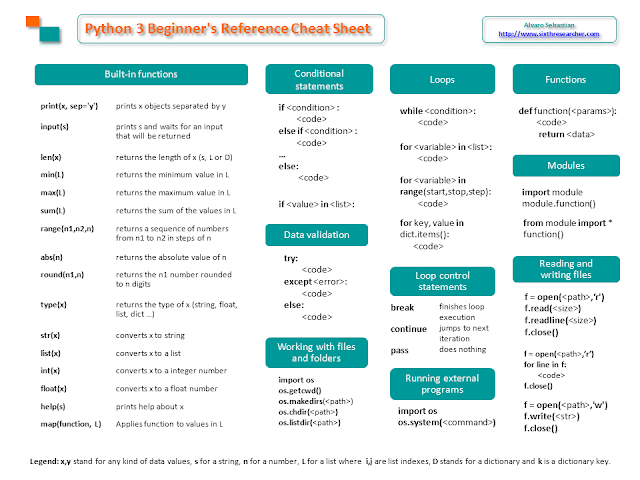
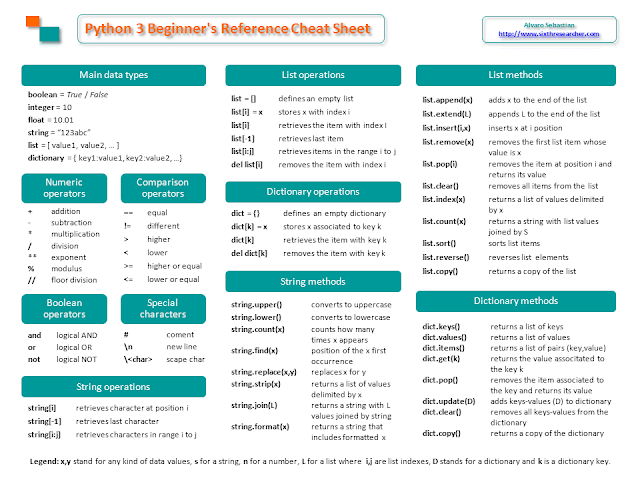
Hola,
cuando intercambiamos datos en al laboratorio a menudo usamos libros Excel, en formato
XLSX, con varias hojas. Ahora bien, si luego queremos convertirlos a ficheros con valores separados por tabuladores (TSV) o comas (CSV), MS Excel sólo te permite hacerlo de hoja en hoja, lo cual es un poco latoso para libros gordos. Aquí os pongo un script en Perl para hacer esta tarea desde el terminal, que requiere instalar el módulo
Spreadsheet::ParseXLSX, por ejemplo con:
$ sudo cpan -i Spreadsheet::ParseXLSX
El código es:
#!/usr/bin/perl -w
# get separate tab-separated (TSV) files from sheets in Excel .xlsx file
use strict;
use Spreadsheet::ParseXLSX;
die "# usage: $0 <infile.xlsx> \n" if(!$ARGV[0]);
my $parser = Spreadsheet::ParseXLSX->new();
my $book = $parser->parse($ARGV[0]);
foreach my $sheet (@{$book->{Worksheet}})
{
open(TSV,'>',"$sheet->{'Name'}.tsv") ||
die "# cannot create $sheet->{'Name'}.tsv: $!\n";
foreach my $row ($sheet->{'MinRow'} .. $sheet->{'MaxRow'})
{
foreach my $col ($sheet->{'MinCol'} .. $sheet->{'MaxCol'})
{
print TSV "$sheet->{'Cells'}->[$row]->[$col]->{'Val'}\t";
} print TSV "\n";
}
close(TSV);
}
Si guarmos el código en un fichero de nombre
xlsx2multitab.pl, podemos invocarlo de la siguiente manera:
$ perl xlsx2multitab.pl libro.xlsx
En el directorio actual se guardará un fichero .tsv por cada hoja del libro,
hasta luego,
Bruno