Hola,
hoy comparto un artículo publicado hace unos meses por Jianbo Xie y colaboradores donde explican su estrategia para anotar pseudogenes en genomas de plantas.
Puedes ver la definición completa de pseudogen en wikipedia, yo la resumo así:
Un pseudogén es un segmento de ADN que deriva de otro gen y que ha perdido al menos parte de su función original en cuanto a su expresión o la codificación de una proteína por acumulación de mutaciones. Se generan por recombinaciones imprecisas, duplicaciones o retrotransposición y en principio pueden confundirse con neogenes al inicio de su ciclo.
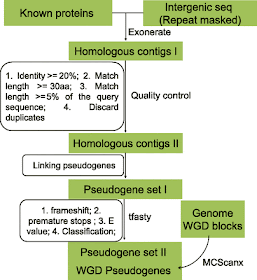
El algoritmo de Xie et al para identificar pseudogenes en zonas intergénicas enmascaradas, donde se supone que no hay genes, se resume en este diagrama:
 |
| http://www.plantcell.org/content/31/3/563.long |
Como podéis ver utiliza exonerate y tfasty (más preciso) para alinear secuencias de proteínas conocidas contra ADN genómico, BLASTP y Orthomcl para grupar los pseudogenes en familias, y finalmente MCScanX para localizar parejas de pseudogenes posiblemente relacioanas por duplicación.
Un saludo,
Bruno
En otra charla hoy he escuchado a José Pérez Silva (https://scholar.google.es/citations?user=rp8yUJMAAAAJ) hablar de un proyecto de aprendizaje automático (Phinder) para reconocer pseudogenes en base al número de alelos, %gc, núm exones, longitud de los exones y de los intrones
ResponderEliminar