sin duda BLAST es una de las herramientas esenciales para cualquiera que se dedique a la bioinformática. En una entrada anterior ya comentábamos que actualmente está vigente la rama BLAST+ del software, que ahora mismo va por la versión 2.2.28, con nuevas funcionalidades y una interfaz más fácil de usar. Sin embargo, como comentan algunos usuarios de SEQanswers o Biostar, las versiones actuales de BLAST no permiten aprovechar al máximo la capacidad de los actuales procesadores multicore, ya que sólo parte de la búsqueda de secuencias similares está paralelizada en el código. Esta es posiblemente una de las razones por las que han sido tan habituales los clusters de cálculo en los laboratorios de bioinformática, que sí permiten hacer en paralelo este tipo de cálculos. Estos son algunos ejemplos de artículos que discuten este tema: http://bioinformatics.oxfordjournals.org/content/19/14/1865 ,
http://bioinformatics.oxfordjournals.org/content/27/2/182 ,
http://www.korilog.com/index.php/KLAST-high-performance-sequence-similarity-search-tool.html .
A lo que iba, ahora mismo cualquiera tiene delante una máquina multicore (en Linux comprueba /proc/cpuinfo) y con ella podremos acelerar significativamente nuestras búsquedas con BLAST si se cumple una condición:
1) la memoria RAM de tu hardware debe superar con creces el tamaño de la base de secuencias que queremos rastrear
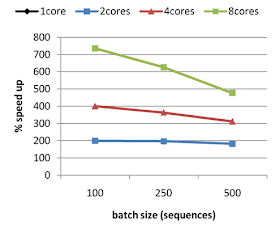
Si esta condición se cumple en tu caso, sigue leyendo. El siguiente código Perl, que gestiona procesos con fork por medio del módulo Parallel-ForkManager, te permitirá exprimir tú máquina, partiendo el problema inicial en pedazos de tamaño igual que serán enviados de manera eficiente a procesar a los cores disponibles de tu máquina. En mis pruebas, el tamaño óptimo de pedazo es de 100 secuencias, y el número de cores óptimo es el físico de la máquina en cuestión.
El script se ejecuta modificando el comando nativo de BLAST, por ejemplo con 8 cores y trabajos de 250 secuencias:
$ split_blast.pl 8 250 blastp -query input.faa -db uniprot.fasta -outfmt 6
Para más detalles guarda el código en un archivo y ejecútalo en el terminal:
#!/usr/bin/perl -w
# Mar2013 Bruno Contreras http://www.eead.csic.es/compbio/
# Script to take advantage of multicore machines when running BLAST,
# which seems to scale up to the number of physical cores in our tests.
# Supports old BLAST (blastall) and new BLAST+ binaries.
#
# The gain in speed is proportional to the number of physical cores allocated,
# at the cost of proportionaly increasing the allocated memory. For this reason probably is not
# a good idea to use it in searches against huge databases such as nr or nt, unless your RAM allows it.
#
# Requires module Parallel::ForkManager (http://search.cpan.org/perldoc?Parallel%3A%3AForkManager)
use strict;
if(eval{ require Parallel::ForkManager }){ import Parallel::ForkManager; }
else
{
die "\n# Failed to locale required module Parallel::ForkManager\n".
"# Please install it by running in your terminal (as a root, preferably with sudo):\n\$ cpan -i Parallel::ForkManager\n\n";
}
my $VERBOSE = 1;
my $DEFAULTBATCHSIZE = 100;
my $BLASTDBVARNAME = 'BLASTDB';
# constant as indices to access read_FASTA_file_arrays
use constant NAME => 0;
use constant SEQ => 1;
my ($n_of_cores,$batchsize,$raw_command,$command) = (1,$DEFAULTBATCHSIZE,'','');
my ($outfileOK,$blastplusOK,$input_seqfile,$output_file,$db_file) = (0,0,'','','');
my $start_time;
if(!$ARGV[2])
{
print "\nScript to take advantage of multicore machines when running BLAST.\n\n";
print "Usage: perl split_blast.pl <number of processors/cores> <batch size> <blast command>\n\n";
print "<number of processors/cores> : while 1 is accepted, at least 2 should be requested\n";
print "<batch size> : is the number of sequences to be blasted in each batch, $DEFAULTBATCHSIZE works well in our tests\n";
print "<blast command> : is the explicit blast command that you would run in your terminal, either BLAST+ or old BLAST\n\n";
print "Example: split_blast.pl 8 250 blastp -query input.faa -db uniprot.fasta -outfmt 6 -out out.blast\n\n";
print "Please escape any quotes in your BLAST command. For instance:\n\n";
print "blastall -p blastp -i input.faa -d nr.fasta -m 8 -F 'm S' -o out.blast\n\n";
print "should be escaped like this:\n\n";
die "blastall -p blastp -i input.faa -d nr.fasta -m 8 -F \\'m\\ S\\' -o out.blast\n";
}
else ## parse command-line arguments
{
$n_of_cores = shift(@ARGV);
if($n_of_cores < 0){ $n_of_cores = 1 }
$batchsize = shift(@ARGV);
if($batchsize < 0){ $batchsize = $DEFAULTBATCHSIZE } # optimal in our tests
$raw_command = join(' ',@ARGV);
if($raw_command =~ /\-i (\S+)/)
{
$input_seqfile = $1;
if($raw_command =~ /\-o (\S+)/){ $output_file = $1 }
if($raw_command =~ /\-d (\S+)/){ $db_file = $1 }
}
elsif($raw_command =~ /\-query (\S+)/)
{
$blastplusOK = 1;
$input_seqfile = $1;
if($raw_command =~ /\-out (\S+)/){ $output_file = $1 }
if($raw_command =~ /\-db (\S+)/){ $db_file = $1 }
}
else{ die "# ERROR: BLAST command must include an input file [-i,-query]\n" }
if(!-s $input_seqfile){ die "# ERROR: cannot find input file $input_seqfile\n" }
elsif(!-s $db_file &&
($ENV{$BLASTDBVARNAME} && !-s $ENV{$BLASTDBVARNAME}.'/'.$db_file) )
{
die "# ERROR: cannot find BLAST database file $db_file\n"
}
# remove BLAST threads commands
$command = $raw_command;
$command =~ s/\-num_threads \d+//;
$command =~ s/\-a \d+//;
if(!$output_file)
{
import File::Temp qw/tempfile/;
my ($fh, $filename) = tempfile();
$output_file = $filename;
if($blastplusOK){ $command .= " -out $output_file " }
else{ $command .= " -o $output_file " } #print "$command\n";
}
else{ $outfileOK = 1 }
print "# parameters: max number of processors=$n_of_cores ".
"batchsize=$batchsize \$VERBOSE=$VERBOSE\n";
print "# raw BLAST command: $raw_command\n\n";
}
if($outfileOK)
{
use Benchmark;
$start_time = new Benchmark();
}
## parse sequences
my $fasta_ref = read_FASTA_file_array($input_seqfile);
if(!@$fasta_ref)
{
die "# ERROR: could not extract sequences from file $input_seqfile\n";
}
## split input in batches of max $BLASTBATCHSIZE sequences
my ($batch,$batch_command,$fasta_file,$blast_file,@batches,@tmpfiles);
my $lastseq = scalar(@$fasta_ref)-1;
my $total_seqs_batch = $batch = 0;
$fasta_file = $input_seqfile . $batch;
$blast_file = $input_seqfile . $batch . '.blast';
$batch_command = $command;
if($batch_command =~ /\-i (\S+)/){ $batch_command =~ s/-i \Q$1\E/-i $fasta_file/ }
if($batch_command =~ /\-query (\S+)/){ $batch_command =~ s/-query \Q$1\E/-query $fasta_file/ }
$batch_command =~ s/$output_file/$blast_file/;
push(@batches,$batch_command);
push(@tmpfiles,[$fasta_file,$blast_file,$batch_command]);
open(BATCH,">$fasta_file") || die "# EXIT : cannot create batch file $fasta_file : $!\n";
foreach my $seq (0 .. $#{$fasta_ref})
{
$total_seqs_batch++;
print BATCH ">$fasta_ref->[$seq][NAME]\n$fasta_ref->[$seq][SEQ]\n";
if($seq == $lastseq || ($batchsize && $total_seqs_batch == $batchsize))
{
close(BATCH);
if($seq < $lastseq) # take care of remaining sequences/batches
{
$total_seqs_batch = 0;
$batch++;
$fasta_file = $input_seqfile . $batch;
$blast_file = $input_seqfile . $batch . '.blast';
$batch_command = $command;
if($batch_command =~ /\-i (\S+)/){ $batch_command =~ s/-i \Q$1\E/-i $fasta_file/ }
if($batch_command =~ /\-query (\S+)/){ $batch_command =~ s/-query \Q$1\E/-query $fasta_file/ }
$batch_command =~ s/$output_file/$blast_file/;
push(@batches,$batch_command);
push(@tmpfiles,[$fasta_file,$blast_file,$batch_command]);
open(BATCH,">$fasta_file") ||
die "# ERROR : cannot create batch file $fasta_file : $!\n";
}
}
}
undef($fasta_ref);
## create requested number of threads
if($batch < $n_of_cores)
{
$n_of_cores = $batch;
print "# WARNING: using only $n_of_cores cores\n";
}
my $pm = Parallel::ForkManager->new($n_of_cores);
## submit batches to allocated threads
foreach $batch_command (@batches)
{
$pm->start($batch_command) and next; # fork
print "# running $batch_command in child process $$\n" if($VERBOSE);
open(BATCHJOB,"$batch_command 2>&1 |");
while(<BATCHJOB>)
{
if(/Error/){ print; last }
elsif($VERBOSE){ print }
}
close(BATCHJOB);
if($VERBOSE)
{
printf("# memory footprint of child process %d is %s\n",
$$,calc_memory_footprint($$));
}
$pm->finish(); # exit the child process
}
$pm->wait_all_children();
## put together BLAST results, no sort needed
my $blastOK = 1;
unlink($output_file) if(-s $output_file && $outfileOK);
foreach my $file (@tmpfiles)
{
($fasta_file,$blast_file,$batch_command) = @$file;
if(!-e $blast_file)
{
unlink($output_file) if(-e $output_file);
if($blastOK)
{
print "# ERROR : did not produced BLAST file $blast_file ,".
" probably job failed: $batch_command\n";
}
$blastOK = 0;
}
else
{
print "# adding $blast_file results to $output_file\n" if($VERBOSE);
system("cat $blast_file >> $output_file"); # efficient, less portable
}
unlink($fasta_file,$blast_file);
}
exit if(!$blastOK);
if(!$outfileOK)
{
open(OUTBLAST,$output_file) || die "# ERROR : cannot read temporary outfile $output_file : $!\n";
while(<OUTBLAST>)
{
print;
}
close(OUTBLAST);
unlink($output_file);
}
else
{
my $end_time = new Benchmark();
print "\n# runtime: ".timestr(timediff($end_time,$start_time),'all')."\n";
}
if($VERBOSE)
{
printf("# memory footprint of parent process %d is %s\n",
$$,calc_memory_footprint($$));
}
####################################################
sub read_FASTA_file_array
{
# in FASTA format
# returns a reference to a 2D array for 4 secondary keys: NAME,SEQ
# first valid index (first sequence) is '0'
my ( $infile ) = @_;
my (@FASTA,$name,$seq,$n_of_sequences);
$n_of_sequences = -1;
open(FASTA,"<$infile") || die "# read_FASTA_sequence: cannot read $infile $!:\n";
while(<FASTA>)
{
next if(/^$/);
next if(/^#/);
if(/^\>(.*?)[\n\r]/)
{
$n_of_sequences++; # first sequence ID is 0
$name = $1; #print "# $n_of_sequences $name\n";
$FASTA[$n_of_sequences][NAME] = $name;
}
elsif($n_of_sequences>-1)
{
$_ =~ s/[\s|\n|\-|\.]//g;
$FASTA[$n_of_sequences][SEQ] .= $_; # conserve case
}
}
close(FASTA);
return \@FASTA;
}
sub calc_memory_footprint
{
my ($pid) = @_;
my $KB = 0;
my $ps = `ps -o rss $pid 2>&1`;
while($ps =~ /(\d+)/g){ $KB += $1 }
return sprintf("%3.1f MB",$KB/1024);
}
Si prefieres hacerlo en Python prueba estas soluciones:
http://qiime.org/scripts/parallel_blast.html ,
http://www.ruffus.org.uk/examples/bioinformatics/
Hasta luego,
Bruno
Actualización 17/04/2013
Unos días después de compartir este código descubro GNU parallel (http://www.gnu.org/software/parallel), todavía no instalado por defecto en los Linux que tengo a mi alcance, que generaliza y simplifica hasta el extremo la tarea que hacíamos con Perl, con algo como:
$ cat input.faa | parallel --block 100k --recstart '>' --pipe blastp -outfmt 6 -db uniprot.fasta -query -
Estos dos comandos concatenados por una tubería 1) parten el archivo de secuencias en bloques de 100Kbytes, separando las entradas por medio del carácter '>', y 2) envían cada bloque a CPUs/cores disponibles en ese momento. Esencialmente es lo mismo que hace el código Perl. En mis pruebas tiene una ganancia en tiempo de cálculo muy similar, con un consumo de memoria también similar.
Solo una pequeña aclaración: el módulo Parallel::ForkManager no gestiona hebras, sino que crea nuevos procesos mediante fork(2).
ResponderEliminarhttps://es.wikipedia.org/wiki/Hilo_de_ejecuci%C3%B3n
https://es.wikipedia.org/wiki/Bifurcaci%C3%B3n_%28sistema_operativo%29
Hola,
ResponderEliminarhe modificado expresiones regulares que causaban problemas con nombres de archivos con caracteres especiales,
añadiendo los modificadores \Q y \E para 'escaparlos':
if($batch_command =~ /\-i (\S+)/){ $batch_command =~ s/-i $1/-i $fasta_file/ }
if($batch_command =~ /\-query (\S+)/){ $batch_command =~ s/-query $1/-query $fasta_file/ }
Un saludo,
Bruno